9767. Понк
Уоршалл
Слушание рок-музыки меняет
вашу ядерную ДНК. Этот поразительный и невероятный факт был недавно опубликован
в Rock Nature Weekly, одном из ведущих научных журналов на планете. Частью
исследования было взятие образцов ДНК у добровольцев как до, так и после сезона
рок-концертов. Образцы были обработаны, и из них были выделены различные гены.
Для каждого человека каждый ген был выделен дважды: вариант до рок-сезона и
вариант после сезона. Эти два варианта были парными, и во многих случаях было
обнаружено, что один вариант представляет собой некоторую перестановку другого
варианта в паре.
Следующим шагом в
исследовании является определение того, как происходят перестановки.
Преобладающая гипотеза предполагает, что перестановка состоит из
последовательности транспозиций, так называемых обменов. Обмен – это событие (его химия еще
полностью не изучена), при котором ровно два азотистых основания в гене
меняются местами. Никакие другие азотистые основания гена обменом не
затрагиваются. Положения двух замененных нуклеотидных оснований могут быть
совершенно произвольными.
Чтобы предсказать и
наблюдать движение молекул в процессе перестановки, исследователям необходимо
знать теоретическое минимальное количество перестановок, которые могут привести
к конкретной перестановке азотистых оснований в гене. Напоминаем, что ген
ядерной ДНК представляет собой последовательность азотистых оснований цитозина,
гуанина, аденина и тимина, которые кодируются как C, G, A и T соответственно.
Вход.
Содержит две строки. Каждая строка содержит n заглавных букв “A”, “C”, “G” или “T” (1 ≤
n ≤ 106). Две строки
представляют собой одну пару некоторых версий гена. Первая строка задает ген до
рок-сезона, вторая строка задает тот же ген от того же человека после
рок-сезона. Число вхождений каждого азотистого основания одинаково в обеих
строках.
Выход.
Выведите минимальное количество обменов, преобразующих первую версию гена во
вторую.
|
Пример входа 1 |
Пример выхода 1 |
|
CGATA ATAGC |
2 |
|
Пример входа 2 |
Пример выхода 2 |
|
CTAGAGTCTA TACCGTATAG |
7 |
РЕШЕНИЕ
графы -
циклы
Анализ алгоритма
Построим граф из
4 вершин: A, C, T, G. Пусть dna1 и dna2 – входные строки. Нам следует при помощи обменов
преобразовать dna1[i] в dna2[i]. Проведем в графе мультиребра: для каждого i (0 ≤ i <
n) проведем
ориентированное ребро (dna1[i], dna2[i]).
Пусть ecnt[u][v]
содержит количество ориентированных ребер u → v. Далее посчитаем количество циклов длины 2, 3 и 4 графе.
Поскольку граф состоит из 4 вершин, то он может содержать цикл максимум из 4
вершин.
Количество
циклов длины 2 между вершинами u и v равно r = min(ecnt[u][v], ecnt[v][u]). Удалим
эти циклы из графа. Для этого следует вычесть r из ecnt[u][v] и ecnt[v][u]. Для
каждого цикла длины 2 достаточно совершить 1 обмен.

Для нахождения циклов
длины 3 следует перебрать тройки вершин (u, v,
w). Их количество
равно r
= min(ecnt[u][v], ecnt[v][w], ecnt[w][u]). Снова удаляем
эти циклы из графа, вычитая r из ecnt[u][v], ecnt[v][w],
ecnt[w][u]. Для каждого цикла длины 3 достаточно совершить 2 обмена.
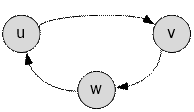
После удаления
циклов длины 2 и 3 в графе останутся только циклы длины 4. Подсчитаем сумму s всех
оставшихся значений ecnt[u][v] по всем
парам (u, v). Полученная сумма s будет
кратна 4. Для каждого цикла длины 4 следует совершить 3 обмена.
Пример
Граф из первого
примера имеет следующий вид. Он соответствует преобразованию, приведенному
справа.
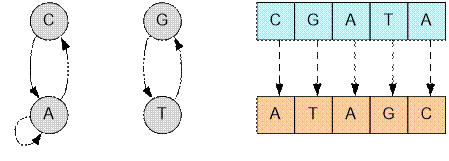
Граф содержит
два цикла длины 2. Следовательно для преобразования первой строки во вторую
достаточно 2 обмена (по одному обмену на каждый цикл).
Построим граф
для второго примера.
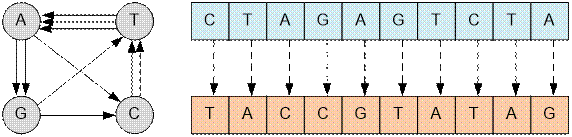
Граф, например,
содержит два цикла длины 3:
·
A
→ G → T;
·
A
→ C → T;
Для уничтожения
каждого из них следует провести два обмена. Удалив эти два цикла, получим граф:
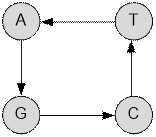
Остался цикл
длины 4, на который следует потратить 3 обмена.
Реализация алгоритма
Объявим матрицу смежности графа ecnt из 4
вершин. Граф
допускает мультиребра. Значение ecnt[u][v] содержит количество
ориентированных ребер u → v. При
помощи отображения letterid каждой
букве поставим в соответствие номер вершины графа.
vector<vector<int>> ecnt(4, vector<int>(4));
map<char, int> letterid{ {'A', 0}, {'C', 1}, {'G', 2}, {'T', 3} };
Читаем входные строки.
cin >> dna1 >> dna2;
Строим граф. Из dna1[i] в dna2[i] проводим
ориентированное ребро.
len = dna1.length();
for (i = 0;
i < len; ++i)
{
l1 = letterid[dna1[i]];
l2 = letterid[dna2[i]];
ecnt[l1][l2]++;
}
В переменной result подсчиываем
количество обменов.
result = 0;
Подсчитываем количество циклов длины 2 и удаляем их из
матрицы смежности.
for (i = 0;
i < 4; i++)
for (j = i
+ 1; j < 4; j++)
{
mn = min(ecnt[i][j], ecnt[j][i]);
result += mn;
ecnt[i][j] -= mn;
ecnt[j][i] -= mn;
}
Подсчитываем количество циклов длины 3 и удаляем их из
матрицы смежности.
for (i = 0;
i < 4; i++)
for (j = 0;
j < 4; j++)
for (k = 0;
k < 4; k++)
{
if (i == j || i == k || j == k) continue;
mn =
min(min(ecnt[i][j], ecnt[j][k]), ecnt[k][i]);
result += 2 *
mn;
ecnt[i][j] -= mn;
ecnt[j][k] -= mn;
ecnt[k][i] -= mn;
}
В переменной rest подсчитываем оставшиеся
ребра. Все они принадлежат циклам длины 4.
rest = 0;
for (i = 0;
i < 4; ++i)
for (j = 0;
j < 4; ++j)
if (i != j) rest
+= ecnt[i][j];
rest ребер образуют rest / 4 циклов длины 4. На каждый цикл требуется 3
обмена.
result += 3 * rest / 4;
Выводим ответ.
cout << result << endl;